IRM de Diffusion!
Ceci est une page en construction.
1) Introduction
Bienvenue sur ce tutorial consacré au traitement des données Bruker de diffusion et de haute résolution pour l’analyse de la structure cardiaque.
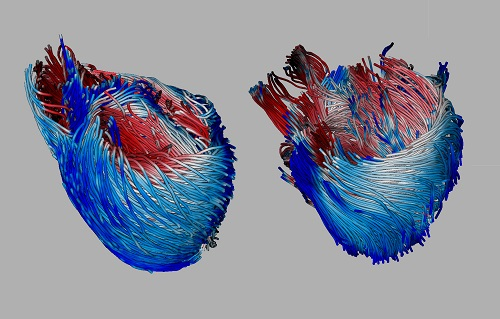
1.1) Sommaire
Les questions ( dans le désordre ) que vous vous posez.
1.2) Les prérequis
Aucun pré-requis n’est nécessaire. Si toutefois ce tutorial est incomplet et/ou contient des erreurs/approximations, n’hésitez pas à nous en faire part. Vous pouvez évidemment contribuer, corriger, et/ou créer très rapidement votre page.
Compter une demi-journée de travail pour traiter votre premier jeu de données. Après quelques jours, 30 minutes suffisent pour tout effectuer.
1.3) (à compléter) Extraction des données de la console Bruker sous format DICOM
Nous allons utliser le logiciel Paravision de Bruker pour extraire les images au format DICOM.
1.4) (à compléter) Copie des données de la console Bruker sous format Bruker
Lors de l’examen, les données Bruker sont stockés dans des dossiers numérotés par ordre d’acquisition des séquences. Par exemple, nous allons extraire le dossier numéro 35 du l’examen intitulé 2016-09-09-examen.
Pour cela, ouvrir le menu puis aller dans le dossier ou ouvrir un terminal et taper la commande suivante:
#se déplacer dans un répertoire
cd /opt/PV6/.../2016-09-09-examen
#taper la commande suivante pour obtenir la liste des dossiers
ls
1.5) Bruker folder structure
Pour chaque acquisition numérotée de 1 à N, nous retrouvons la même arborescence avec:
- un fichier texte
acqpqui contient les paramètres d’acquisition. - un fichier texte
methodqui contient d’autres paramètres d’acquisition. - un fichier binaire
fidqui contient les données brutes ie non reconstruites. - une sous dossier
pdatapourProcessed Data, qui contient les données reconstruites. Attention plusieurs reconstructions peuvent être effectuées à partir des mêmes données. - la liste est non exclusive, pour tout renseignement supplémentaire, ce lien est plus complet.
#toujours à partir du terminal, pour vérifier quelle séquence a été jouée,
cd /opt/PV6/.../2016-09-09-examen
grep -R '##$Method=' 35/Dans le sous-dossier pdata (par ex. 2016-09-09-examen/35/pdata), nous trouvons les sous dossiers numérotées (1,2,3 etc) pour chaque nouvelle reconstruction. Nous serons généralement intéressé par les reconstructions numérotées 1 ou 2 suivant les cas.
Dans chaque dossier de reconstruction (par ex. 2016-09-09-examen/35/pdata/1), il y a:
- un fichier binaire
2dseq. C’est le bloc de données qui contient les images en format 3D ou 4D. Par exemple, les données correspondant au direction de diffusion sont stockées successivement à l’intérieur de ce bloc. Nous verrons plus tard comment les extraire. - un fichier texte
recoqui contient les détails concernant la reconstruction effectuées. Par ex. le champ de vue, le nombre de pixels et la résolution - un fichier texte
visu_parsqui contient d’autres détails concernant la reconstruction effectuées. - la liste est aussi non exclusive, pour tout renseignement supplémentaire, ce lien est plus complet.
1.6) Contribuer à ce tutoriel
2) Quelques pré-requis organisationnels
Avant de commencer quelques conseils. Les données peuvent être copiées sur votre espace personnel de stockage : smb://prenom.nom et utilisées sur le pc de post-traitement de l’équipe imagerie. Il est préférable de ne pas créer de doublons de vos données pour préserver l’espace dsique.
2.1) Création de l’arborescence des données IRM
Les données sont copiées par exemple dans le répertoire /home/pc/Reseau/Imagerie/Auckland/ et sont classées par espèce ou par utilisateur puis par ordre d’acquisition. Elles sont donc numérotées avec l’année, le mois et le jour d’acquisition suivi d’un nom libre de choix pour chaque échantillon.
KadenceControlYear_Month_Day_Study2015_11_14_Heart_12016_05_03_Heart_2
Infarct
SpeciesControlYear_Month_Day_Study_1Year_Month_Day_Study_2
InfarctYear_Month_Day_Study_1
UserControlYear_Month_Day_Study_1Year_Month_Day_Study_2
InfarctYear_Month_Day_Study_1
Dans chaque dossier nous retrouvons les données de diffusion notées et les données de haute résolution si les deux sont présentes.
Control2015_11_14_Heart_130(données de diffusion)4(données de haute-résolution)
2016_05_03_Heart_2
2.2) Création de l’arborescence des données de post-traitées
Pour préserver les données acquises de toute mauvaise manipulation, le travail de post-traitement est effectué dans un nouveau dossier nommé STDT. Il est généré en lançant le script suivant:
cd /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/2015_11_14_Heart_1/
#génération des sous dossier
sh createfolder.shSi ce fichier n’est pas présent vous pouvez facilement le générer en suivant ces commandes:
#génération des sous dossier
cd /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/2015_11_14_Heart_1/
echo '' > createfolder.sh
echo '' >> createfolder.sh
#et enfin
sh createfolder.shAinsi nous disposons de l’arborescence suivante:
KadenceControl2015_11_14_Heart_130(données de diffusion)4(données de haute-résolution)STDT(dossier de post-traitement)ST(données de haute-résolution)DT(données de diffusion)DT_PREPROCESSED_VTI(extraction des données Bruker)MASK(masques binaires après seuillage)
Stat(statistique)FILESPNG
2.3) Les librairies Visualisation Tool Kit (VTK) et Image Tool Kit (ITK)
2.4) Les formats de données
Pour permettre l’intéraction entre différents logiciels, nous allons utiliser de nombreux formats de données.
-
les formats propriétaires
Bruker: les données sont stockés en binaire en 16 bit unsign pour les fid et 32 bit unsign pour les images reconstruites. - le format
VTK: c’est celui ci que nous allons principalement utiliser. Ils comprends plusieurs sous format:.vti: le formatimage data, comprend un en-tête en htlm suivi des données stockées en binaire.vtk: le formatDATASET STRUCTURED_POINTS, comprend un en-tête enASCIIsuivi des données stockées en binaire ou enASCII(ancien format). *
- le format matlab
3) La diffusion
Bibliographie à renseigner. En attendant, une définition à minima:
“L’IRM de diffusion est une technique basée sur l’imagerie par résonance magnétique (IRM). Elle permet de calculer en chaque point de l’image la distribution des directions de diffusion des molécules d’eau. Cette diffusion étant contrainte par les tissus environnants, cette modalité d’imagerie permet d’obtenir indirectement la position, l’orientation et l’anisotropie des structures fibreuses, notamment les faisceaux de matière blanche du cerveau”. Source: wikipedia.
3.1) Localisation des algorithmes/programmes/logiciels.
Plusieurs petits programmes ont été développées pour extraire, calculer et visualiser la structure cardiaque, ce protocole est susceptible d’évoluer et d’être affiné. En particulier la comparaison de multiples écchantillons nécessite quelques nouvelles fonctionnalités non développées à ce jour.
En effectant ces commandes vous trouverez la liste des programmes en développement
#localisation des programmes <a id="logiciels"></a>
cd /home/nelsonleouf/Dev/Vtk/BrukerTools/build/
lsNous nous interesserons particulièrement à ces derniers:
DT_fullC_beta_0.x: programme de traitement données de diffusion.Alignement: programme pour orienter nos données.MakeFigure: programme facilitant la création de figures avec Paraview.
En effet, nous allons utiliser plusieurs logiciels de visualisation dont:
Volview: c’est un logiciel très léger qui permet de visualiser les données en 3D. Téléchargeable ici.ITK-Snap: c’est un logiciel de segmentation. Téléchargeable ici.Seg3D2: c’est aussi un logiciel de segmentation. Téléchargeable ici.Paraview: C’est un logiciel de rendu 3D extrêmement au top. Téléchargeable ici.ImageJ: déjà bien connu.Blender: logiciel de rendu 3D, plus que extrêmement au top, la preuve en images. Téléchargeable ici.
NB: Ils sont tous déjà installés sur le pc de post-traitement de l’équipe imagerie.
3.2) Extraction des données de diffusion
Se déplacer dans le dossier suivant pour accéder à l’executable DT_fullC_beta_0.x
#déplacement
cd /home/nelsonleouf/Dev/Vtk/BrukerTools/build/Afin d’extraire les données, plusieurs argument doivent être transmis au programme, ainsi que trois fichiers texte qui contient des paramètres.
Les arguments sont les suivants:
- argument 1 : lien vers l’espace de stockage
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/ - argument 2 : nom du dossier, par ex.
2015_11_14_Heart_1 - argument 3 : numero d’acquisition, par ex.
30 - argument 4 : mode
1,2,3,4ou0, nous y reviendrons.
Les fichiers texte sont les suivants:
info.txt: renseigne la taille et la résolution des imagesthreshold.txt: renseigne les valeurs pour segmenter l’échantillonthreshold_3layers.txt(optionnel) : renseigne les valeurs pour segmenter l’échantillon plus finementaxis.txt: renseigne les coordonnées de l’axe principal du coeur
Ces trois fichiers sont stockées dans la racine de chaque acquisition, par exemple /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/2015_11_14_Heart_1/30/. Vous regarder s’il existe en tapant la commande
#ouverture du fichier info.txt s'il existe
gedit /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/2015_11_14_Heart_1/30/info.txt &Nous commencerons par uniquement renseigner le fichier info.txt qui à la structure suivante:
- taille de la matrice suivant x
- taille de la matrice suivant y
- taille de la matrice suivant z
- champ de vue suivant x
- champ de vue suivant y
- champ de vue suivant z
- drapeau N4 (mettre 0)
- drapeau Seuillage (mettre 1 ou 3)
- drapeau Rotation (mettre 0, 1, 2 ou 3)
Le drapeau seuillage activé à la valeur 3 va découper l’échantillon en 3 zones selon l’axe z afin de segmenter séparemment ces zones. L’étape de seuillage sera alors plus longue. Si vous souhaitez aller plus vite mettre le drapeau Seuillage à 1. Le drapeau rotation doit être à 0 sauf mention contraire. Il permet de tourner l’échantillon dans l’espace, ce qui peut parfois être nécessaire si l’échantillon n’a pas été placé idéalement dans l’aimant.
Pour extraire ces informations relatives à la taille de la matrice et au champ de vue, vous pouvez ouvrir le fichier visu_par.
#ouverture du fichier visu_par et methode de l'acquisition numero 30
gedit /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/2015_11_14_Heart_1/30/2/visu_pars &
#ligne 20 et 21, vous obtenez la taille de la matrice pour les trois directions:
$VisuCoreSize=( 3 )
200 166 200
#ligne 20 et 21, vous obtenez la taille du champ de vue pour les trois directions:
$VisuCoreExtent=( 3 )
120 100 120#ouverture du fichier info.txt s'il existe
gedit /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/2015_11_14_Heart_1/30/info.txt &Et enfin nous lançons la commande
# déplacement si necessaire
cd /home/pc/Dev/Vtk/BrukerTools/build/
# extraction des données avec la commande à 4 arguments
./DT_fullC_beta_0.x /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/ 2015_11_14_Heart_1/ 30 1Plusieurs messages s’affichent, noter l’enregistrement de nombreux fichiers dans votre dossier STDT/DT/ et en particulier dans le sous dossier DT_PREPROCESSED_VTI.
3.2) Visualisation des données
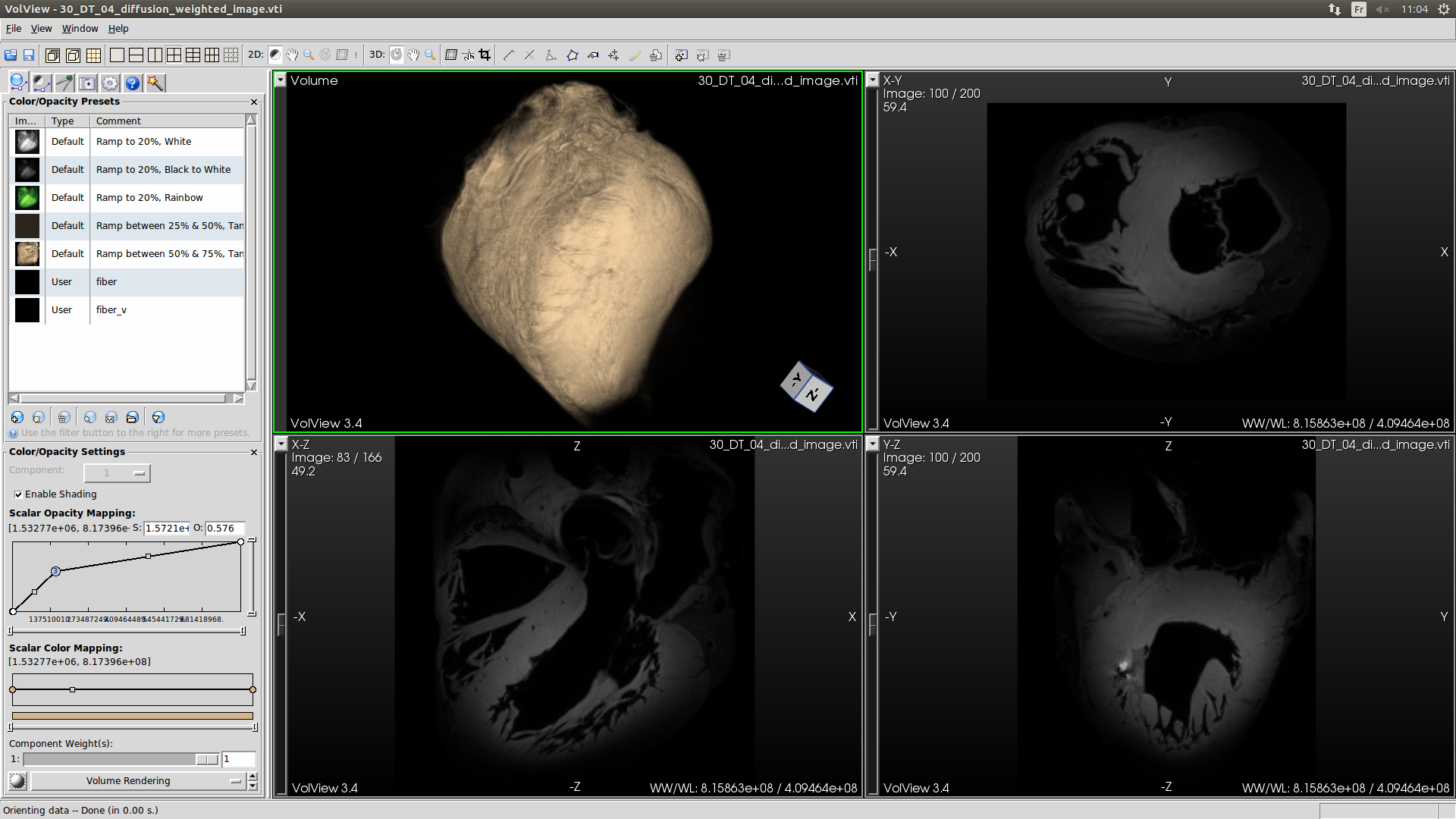
Ouvrez un nouveau terminal et lancer le programme Volview.
# déplacement si necessaire
cd /home/pc/Dev/VolView-3.4-Linux-x86_64/bin/
./VolviewPuis ouvrez le fichier 30_DT_04_diffusion_weighted_image.vti correspondant à l’image pondérée en diffusion. /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/2015_11_14_Heart_1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_04_diffusion_weighted_image.vti.
Puis cliquer sur suivant plusieurs fois.
Autre alternative, ouvrez un nouveau terminal et lancer le programme Itk-snap.
# déplacement si necessaire
cd /home/choupinetleouf/Dev/itksnap-3.4.0-20151130-Linux-x86_64/bin/
./itksnap
3.3) Advanced Normalization Tools (ANTs)
Par la suite, nous allons utliser une librairie nommée ANTs pour Advanced Normalization Tools. ANTs est expliqué plus en détail ici. Quelques remarques.
ANTsvient du monde de la neuro.ANTSest une librairie contenant des fonctions et un ensemble de scripts appelant ces fonctions. Il fonctionne exclusivement en ligne de commande, et permet un traitement complètement automatisé d’un large spectre de données d’imagerie.- Idéalement
ANTsdevrait être utilisé pour traiter nos données. - Nous l’utiliserons pour plusieurs aspects: la correction des inhomogénéités du champs, pour la registration et la création de template.
NB: ANTs est déjà installé sur le pc de post-traitement de l’équipe imagerie.
Sinon cloner ou télécharger le code ici, et on lance ces commandes:
git clone git://github.com/stnava/ANTs.git
mkdir ants-build
cd ants-build
cmake ../ANTs
make -j 8
#puis attendez longtempsRendez-vous sur ANTs pour en savoir plus!
3.4) Correction de biais (N4 ITK bias correcction)
Cette étape est relativement longue (pour l’ordinateur), pour pouvoir passer outre si ce calcul a été fait, un drapeau a été ajouté dans le fichier de configuration info.txt à la ligne treize. 1 code pour effectuer ce calcul, 0 code pour ne pas effectuer ce calcul. Dans un premier temps, nous choisirons d’activer ce calcul en mettant le drapeau à 1.
#ouverture du fichier info.txt et changement de la treizième ligne
gedit /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/2015_11_14_Heart_1/30/info.txt &Nous lançons la commande suivante
# déplacement si necessaire
cd /home/nelsonleouf/Dev/Vtk/DT_fullC_beta_0.x/build/
# lancement de la commande à quatre argument avec le mode n°2
./DT_fullC_beta_0.x /home/pc/Reseau/Imagerie/data-bruker/Espece_2/ coeur_2/ 35 2Une fois ce calcul effectué, vous pouvez regarder avec le logiciel Volview les fichiers résultants et noter les différences en terme d’intensité. Pour cela ouvrez le logiciel Volview comme ceci:
#ouvrer un terminal
cd
cd Dev/Volview/bin
./Volview
#en haut à gauche, cliquer sur menu, puis ouvrir et charger les fichiers suivants:
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/2015_11_14_Heart_1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_04_diffusion_weighted_image.vti
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/2015_11_14_Heart_1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_04_diffusion_weighted_image_cut_N4.vtk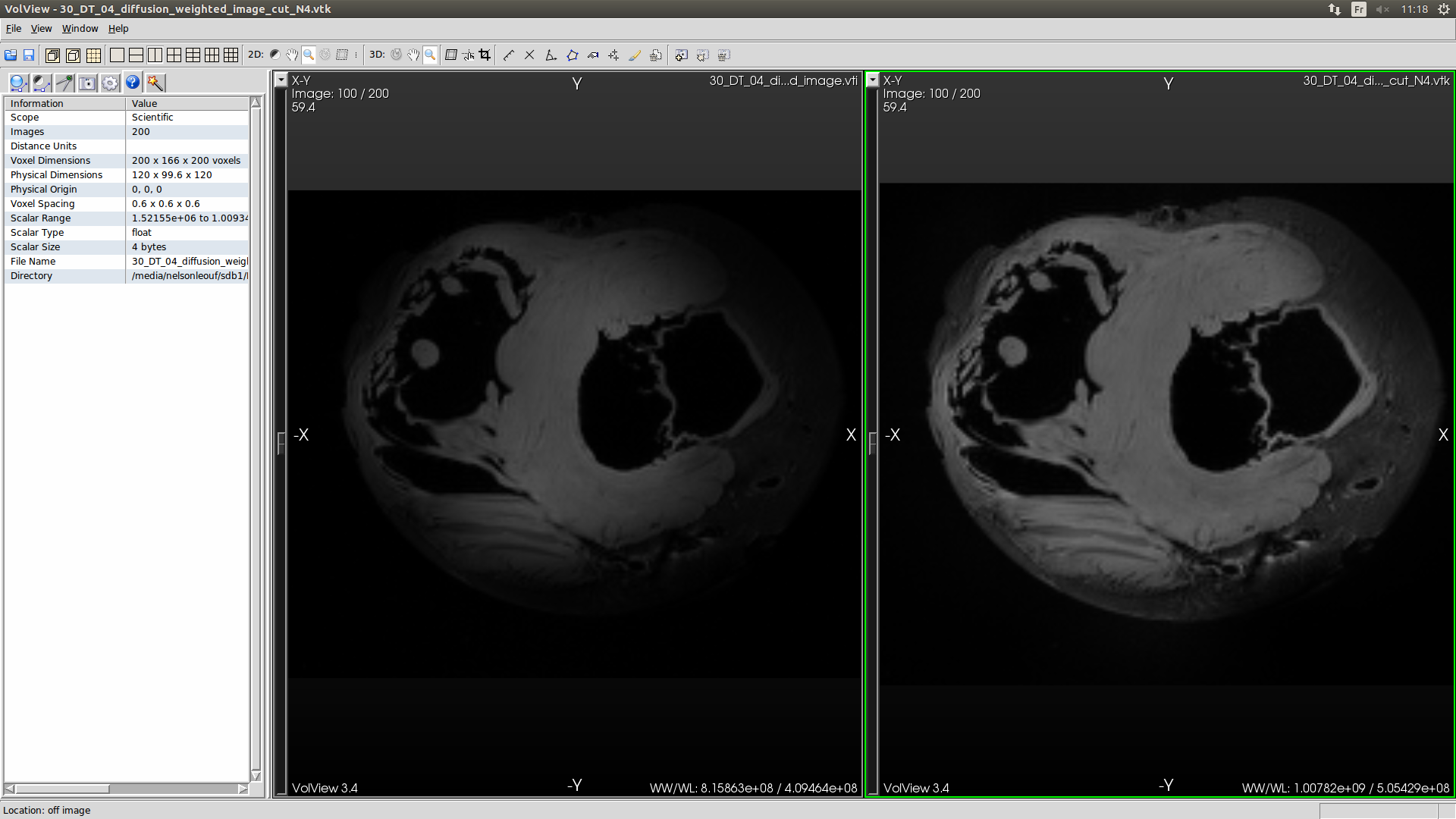
Vous pouvez maintenant désactiver la correction de biais N4 dans le fichier de configuration info.txt en mettant le drapeau à 0.
Discussion scientifique : ceci est coupe short axis…. regarder les différences de contrastes avant et après application du filtre. Modification du contraste global mais pas de mofication à l’échelle de la structure , vérifier la présence de fibre cardiaque visible à l’oeil nu. Noter par ailleurs la présence de graisse autour du ventricule gauche qu’il faudrait segmenter par la suite. La diffusion pas effective sur le tissu graisseux.
3.5) Segmentation
La segmentation peut-être effectuée plus ou moins finement. L’approche suggérée est loin d’être exhaustive mais offre un niveau de précision relativement acceptable pour notre application. Néanmoins de nombreuses altérnatives sont disponibles.
Si le drapeau de seuillage vaut 1, le seuillage est effectué sur tout l’échantillon, s’il vaut 3 l’échantillon est divisé en 3 zones selon l’axe z et le seuillage est effectué séparement sur les 3 zones. Nous allons présenter pas à pas le cas où le drapeau de seuillage vaut 3.
La première étape est un seuillage sur la fraction d’anistropie, la trace et les images pondérée en diffusion. Pour commencer, nous divisons l’échantillon en 3 segments noté apex, mid, et base selon l’axe principal du coeur. Si ce n’est pas le cas veuillez effectuer les rotations nécessaires.
Nous utilisons ensuite le logiciel Seg3D pour déterminer la valeur du seuillage et nous ajoutons alors chaque seuil obtenu dans le fichier de configuration threshold.txt ou threshold_3layers.txt selon les cas. Les valeurs étant sauvegardés dans un fichier texte, il sera alors possible de les modifier pour effectuer des ajustements.
#ouvrez un terminal et déplacez vous vers le répértoire du logiciel de segmentation avec la commande suivante: "cd chemin"
cd Dev/Seg3D2/bin/
./Seg3DClick on Start New Project, choose 2015_11_14_Heart_1_DWI as project name and your personal directory as project path. Then at the top left-hand corner, click on the File, then Import Layer from Single File and open the following file:
#file to open with Seg3D
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/2015_11_14_Heart_1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_01_fractional_anisotropy_gaussian_part1.vtk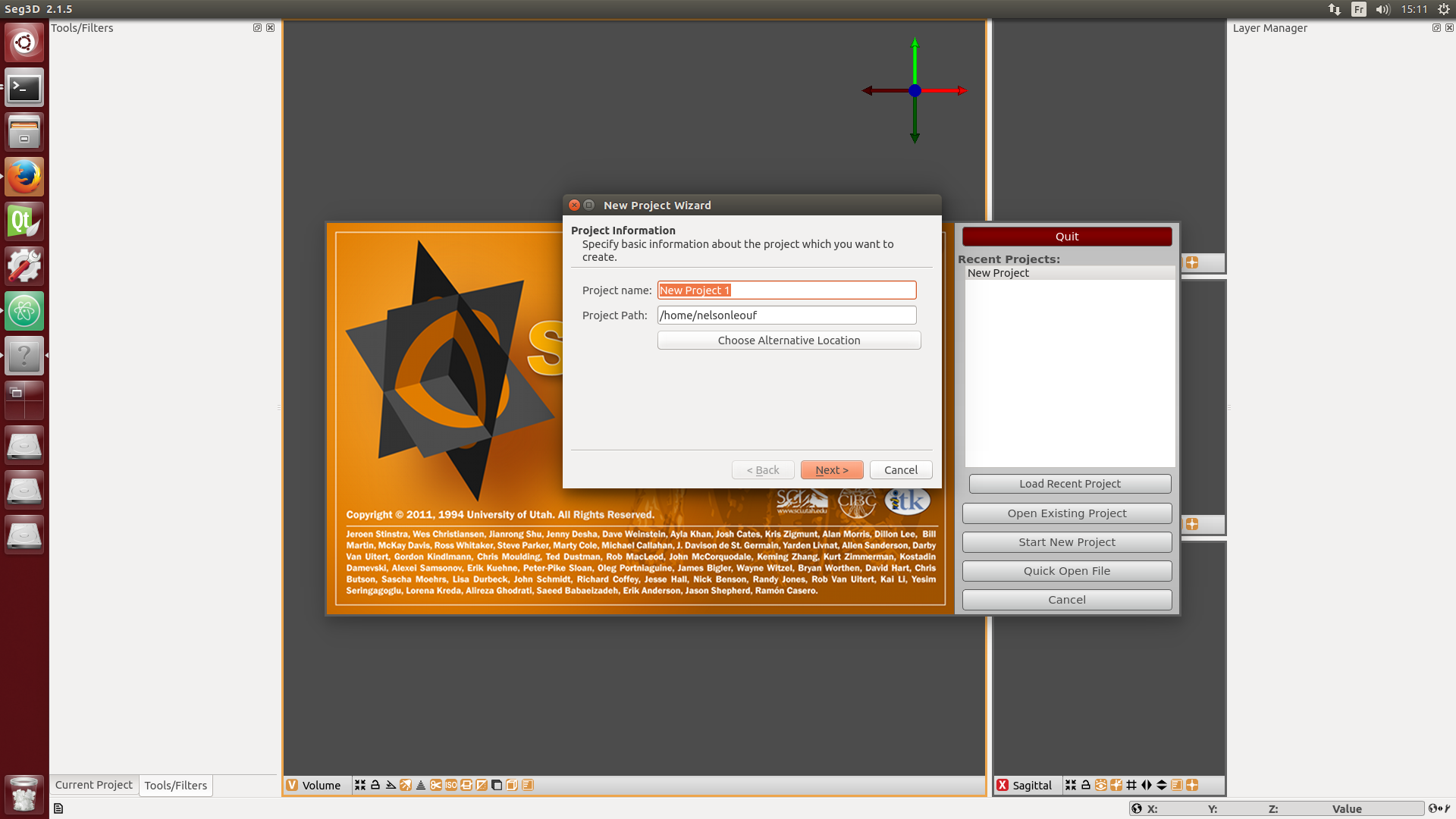
La fenêtre est divisée en 4 sous-fenêtres, si ce n’est pas le cas cliquez sur View puis Two And Two. Vous pouvez maintenant observer votre échantillon et faire défiler les coupes, notons que nous nous situons à l’apex. L’enjeu est de seuiller le plus finement possible. Pour cela, cliquez dans le menu outil et sélectionner l’option Threshold et ajoutez des petites croix sur la zone que vous souhaitez conserver. Ajouter au tant de petits croix que nécessaire en particulier à l’extrémité de l’apex afin de conserver l’anatomie d’origine. Le contraste étant plus faible à cette extrémité, cette zone est facilement oubliée lors de la segmentation.
Aller maintenant dans la rubrique Tools, puis sélectionner l’option Threshold, une fenêtre s’ouvre sur la droite. Cliquer le bandeau Clear Seeds puis cliquer sur l’image sur le tissu que vous souhaitez sélectionner. Une petite croix apparaît répéter cette opération autant que nécessaire. Vous devez obtenir un résultat similaire aux fenêtres suivantes.
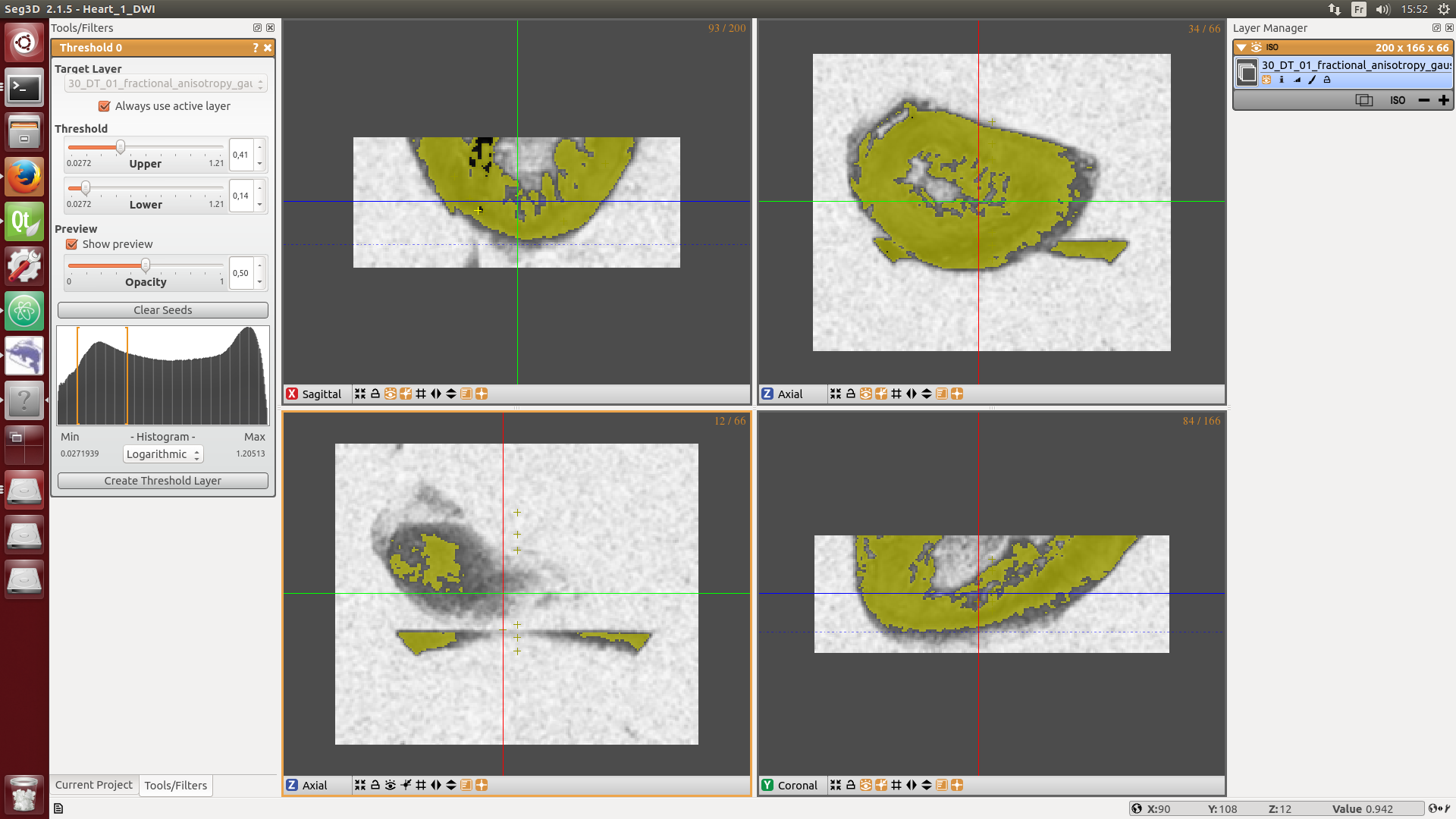
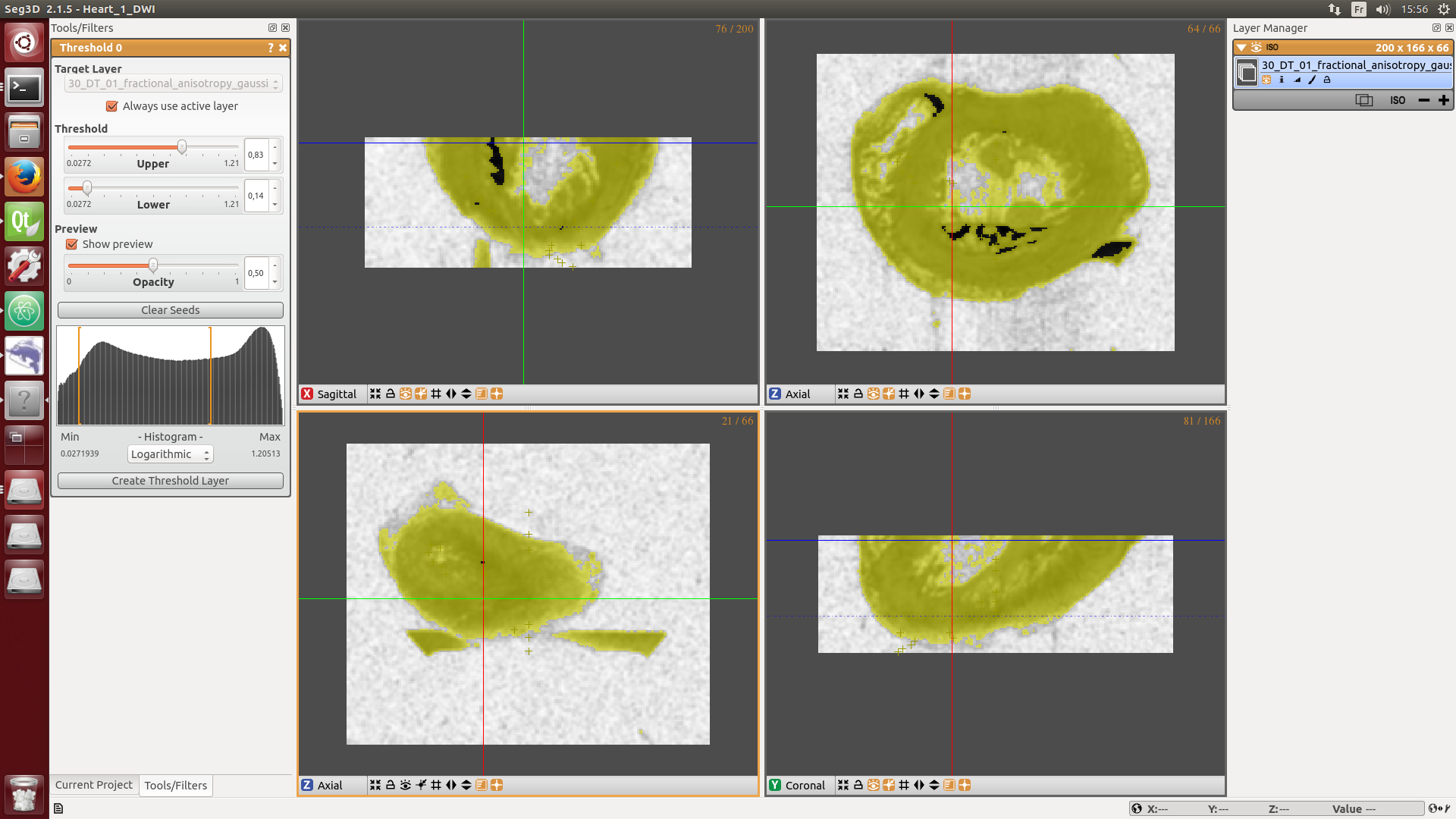
Maintenant notez les deux valeurs (minimales et maximales, ici 0.14 et 0.83) situées dans la fenêtre Upper and Lower sur la première ligne du fichier de configuration threshold_3layers.txt selon cette nomenclature:
FA_min_apex FA_max_apex Trace_min_apex Trace_max_apex DWI_min_apex DWI_max_apex FA_min_mid FA_max_mid Trace_min_mid Trace_max_mid DWI_min_mid DWI_max_mid FA_min_base FA_max_base Trace_min_base Trace_max_base DWI_min_base DWI_max_base
Si le seuillage est réalisé sur tout l’échantillon (le drapeau de seuillage vaut 1), la nomenclature du fichier de configuration threshold.txt est la suivante:
FA_min_apex FA_max_apex Trace_min Trace_max DWI_min DWI_max
Puis passez à la région mid ventriculaire en ouvrant le fichier:
/home/nelsonleouf/DICOM/Heart1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_01_fractional_anisotropy_gaussian_part2.vtkUtilisez l’option Threshold et les petites croix pour segmenter la région mid-ventriculaire. N’hésitez pas enlever la graisse.

Next we’ll add the minimum and maximum values on the second line according to this classification/convention:
FA_min_apexFA_max_apex Trace_min_apex Trace_max_apex DWI_min_apex DWI_max_apex FA_min_mid FA_max_mid Trace_min_mid Trace_max_mid DWI_min_mid DWI_max_mid FA_min_base FA_max_base Trace_min_base Trace_max_base DWI_min_base DWI_max_base
Répèter cette opération pour chaque région et chaque contraste afin de remplir les 24 valeurs, 12 minimales et 12 maximales en chargeant les fichiers suivants.
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_01_fractional_anisotropy_gaussian_part3.vtk
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_01_fractional_anisotropy_gaussian_part1.vtk
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_01_fractional_anisotropy_gaussian_part2.vtk
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_01_fractional_anisotropy_gaussian_part3.vtk
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_04_diffusion_weighted_image_part1.vtk
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_04_diffusion_weighted_image_part2.vtk
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart1/STDTdata/DT/DT_PREPROCESSED_VTI/30_DT_04_diffusion_weighted_image_part3.vtk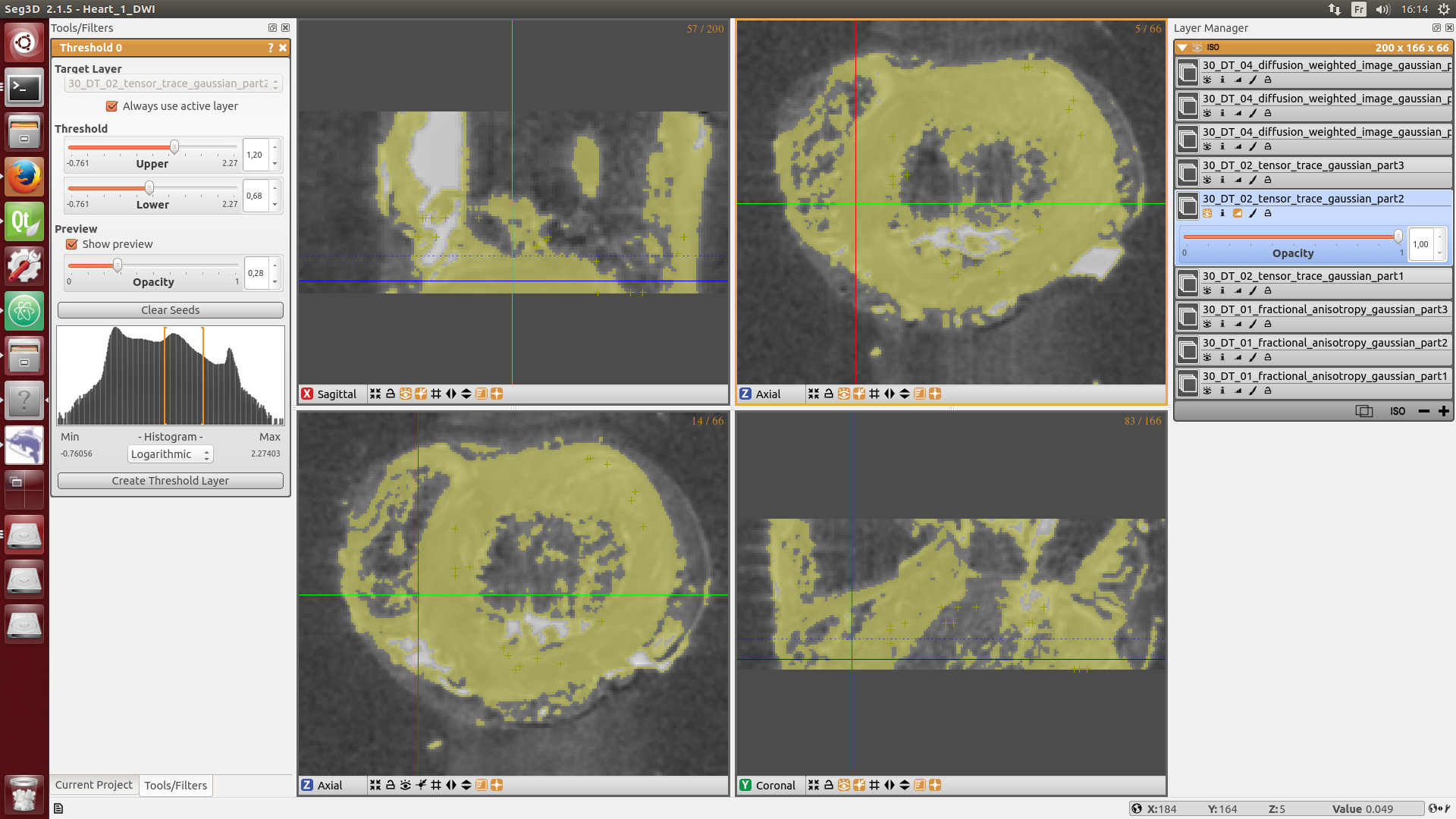
Une fois ces étapes effectuées, les valeurs de segmentation sont sauvegardées, vous obtiendrais un fichier similaire aux lignes ci-dessous. Si la segmentation n’est pas satisfaisante vous pouvez rejouer cette étape autant que nécessaire pour ajuster au mieux les seuillages.
0.23 0.72 0.53 1.14 228580000 794832000
0.19 0.89 0.51 1.10 245244992 778752000
0.20 0.88 0.54 1.06 311708000 966128000
Maintenant, nous sommes prets pour créer notre masque binaire, relancez le programme DT_fullC_beta_0.x avec la commande suivante:
# if necessary, navigate to the build folder using cd
cd /home/nelsonleouf/Dev/Vtk/DT_fullC_beta_0.x/build/
# launch the programme with the following arguments
./DT_fullC_beta_0.x /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/ Heart_1/ 30 2
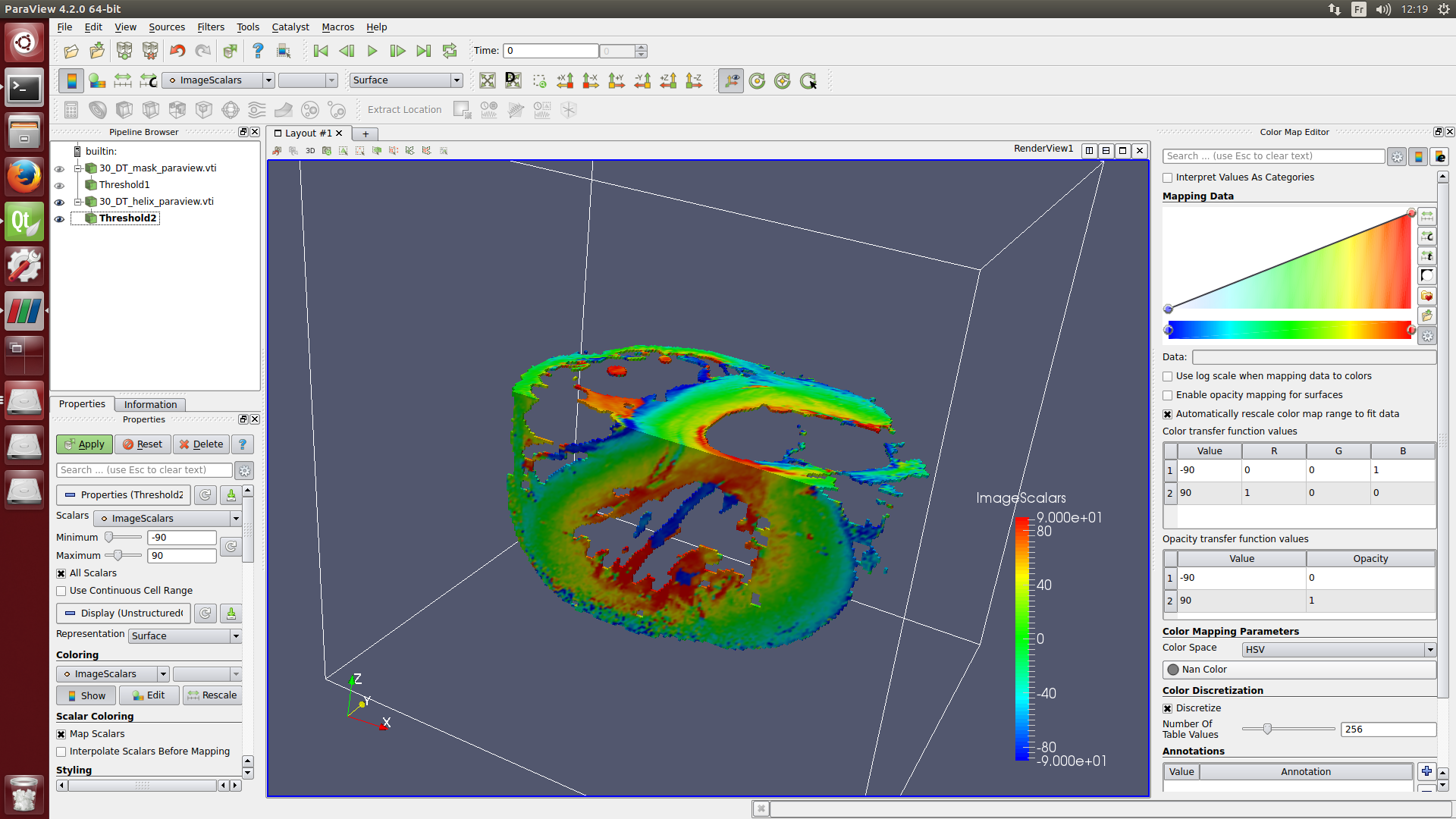
Noter la création de plusieurs masques que vous pouvez ouvrir avec Seg3D ou Volview pour vérifier la qualité de la segmentation:
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart_1//STDTdata/DT/**MASK**/30_DT_mask_3_layers_fractional.vtk
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart_1//STDTdata/DT/**MASK**/30_DT_mask_3_layers_trace.vtk
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart_1//STDTdata/DT/**MASK**/30_DT_mask_3_layers_dwi.vtk
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart_1//STDTdata/DT/**MASK**/30_DT_mask_3_layers_combine.vtk
après utilisation d'un kernel [3,3,3] pour boucher les trous dans le masque binaire
/home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart_1//STDTdata/DT/MASK/30_DT_mask_threshold_3_layers.vtkd) Quelques remarques sur cette étape.
Les volumes que vous venez de charger ont préalablement été filtrés avec un filtre gaussien 3D de kernel [1 1 1] afin d’enlever le bruit présent dans l’image et d’homogénéiser le seuillage.
e) The cardiac coordinate system
3.6)
a) Long axis definition
Nous allons maintenant regarder notre segmentation et définir deux points par lesquel passe l’axe principal du coeur, ces points se situent à l’apex et dans la partie basale du coeur. Nous considérons pour cela uniquement le ventricule gauche Nous ajouterons alors les coordonnées correspondantes dans le fichier de configuration axis.txt.
Pour cela , nous chargeons dans Seg3D le fichier /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart_1//STDTdata/DT/MASK/30_DT_mask_threshold_3_layers_1px.vtk.
Et nous utilisons le curseur, les coordonnées se mettent à jour directement en bas à droite
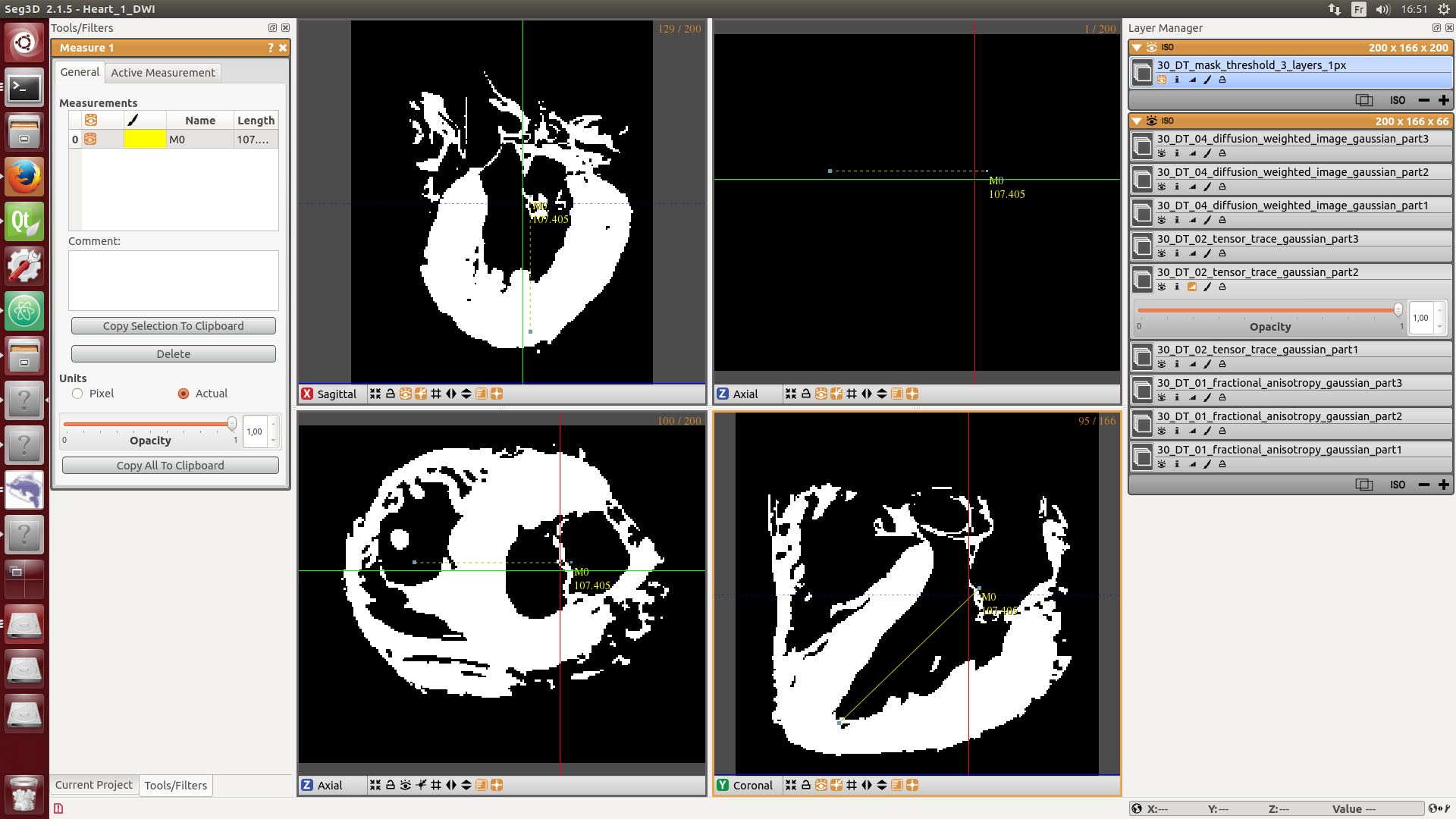
# create and edit the axis.txt file to save the long axis coordinates
nedit /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart_1/30/axis.txt &Maintenant notez les valeurs de x,y,z du fichier selon cette nomenclature:
X_apex Y_apex Z_apex X_base Y_base Z_base
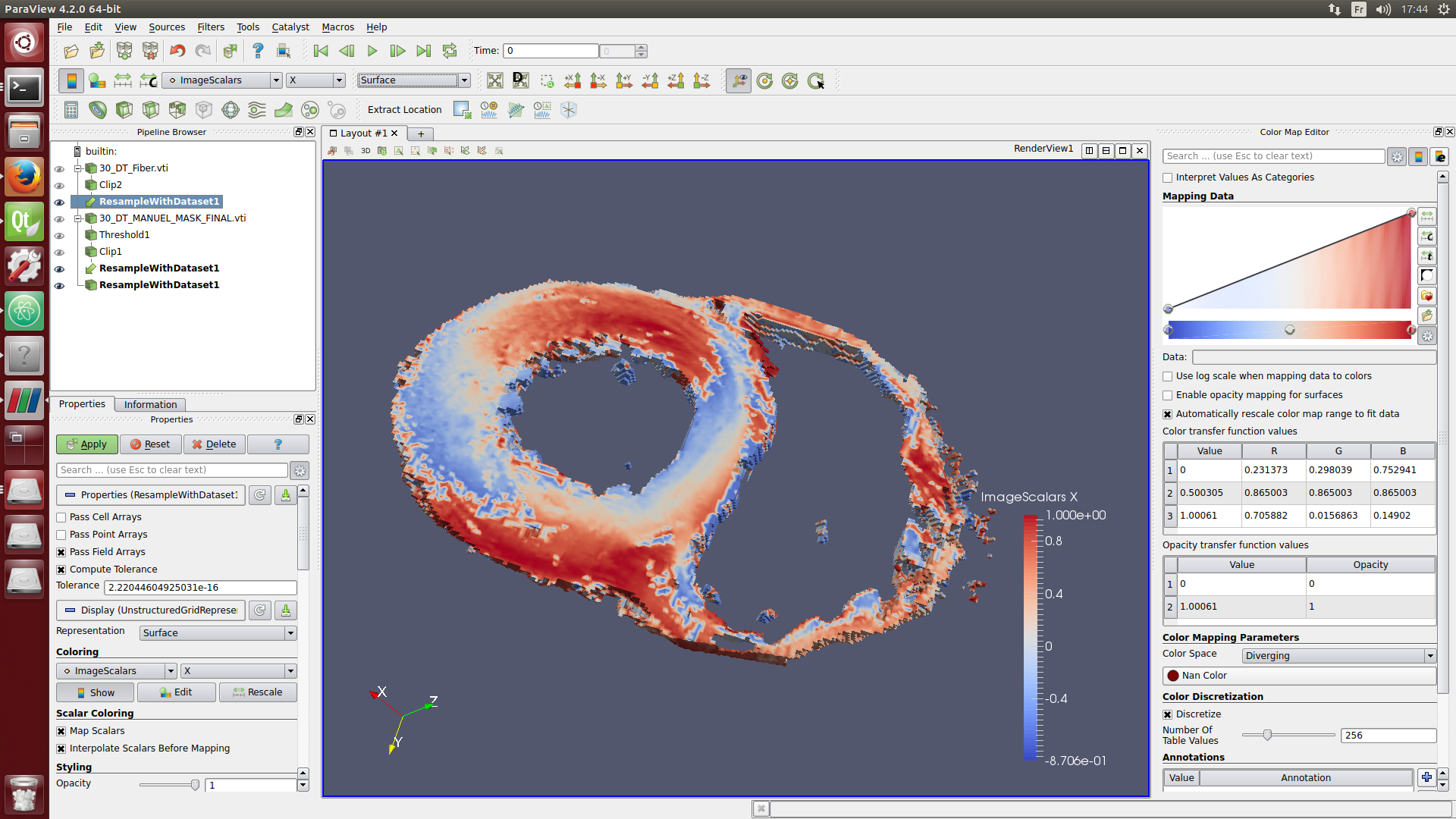
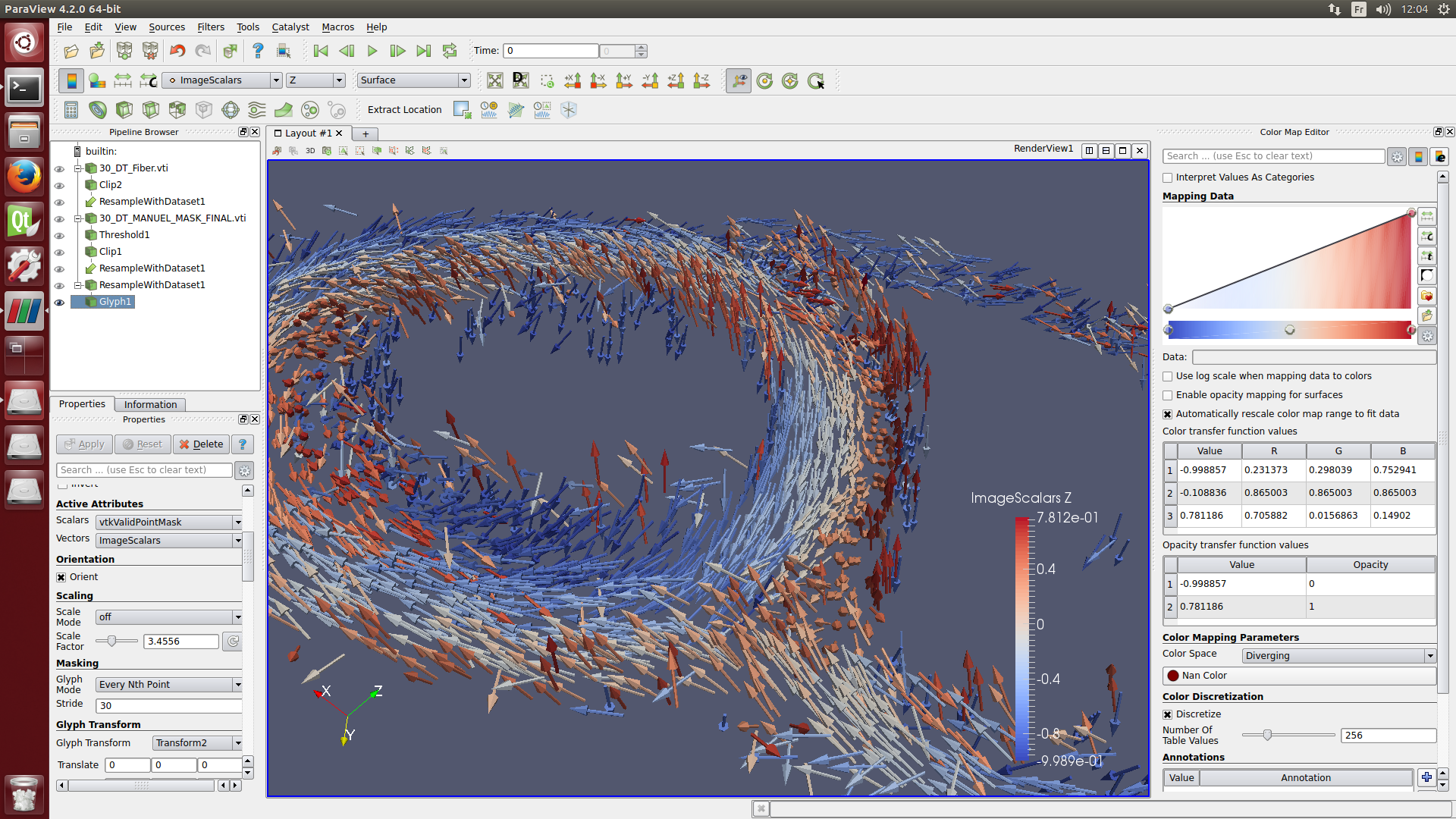
Nous pouvons aussi utiliser le programme Paraview, pour cela ouvrer le fichier /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/Heart_1//STDTdata/DT/MASK/30_DT_mask_threshold_3_layers_1px.vtk dans Paraview.
b) Calcul des angles helix, tranverse, beta …
Il existe plusieurs méthodes pour analyser la microstructure cardiaque. Une des plus courante consiste à projeter les vecteurs propres issus du tenseur de diffusion dans un repère cylindrique puis à calculer l’angle entre la projection du vecteur et les vecteurs directeurs de ce repère. Par exemple l’angle hélix est l’angle entre la projection du premier vecteur propre et l’axe z du repère cylindrique (qui correspond à l’axe principale du coeur). La structure du myocarde est très couramment décrite en utilisant cet angle, en effet en parcourant le myocarde de l’epicarde à l’endocarde, on observe une rotation de l’angle hélix de 120° environ.
#launch the software with 3 as final argument to calculate the helix angle
./DT_fullC_beta_0.x /home/pc/Reseau/Imagerie/Auckland/Kadence/Control/ Heart_1/ 30 34) Réalisation des figures
4.1) Les images de magnitudes
Uiliser le répertoire createMultiExectuble, puis ajouter DTFullBeta0_3 a celui ci avec la librairie et Faire un second executable vtk qui lit l’image de magnitude et qui imprime les images en short axis en png , utiliser un commande bash pour créer le git et compresser le tout.
4.2) L’orientation des fibres
4.3) Superposition
a) Affichage des résultats dans Volview
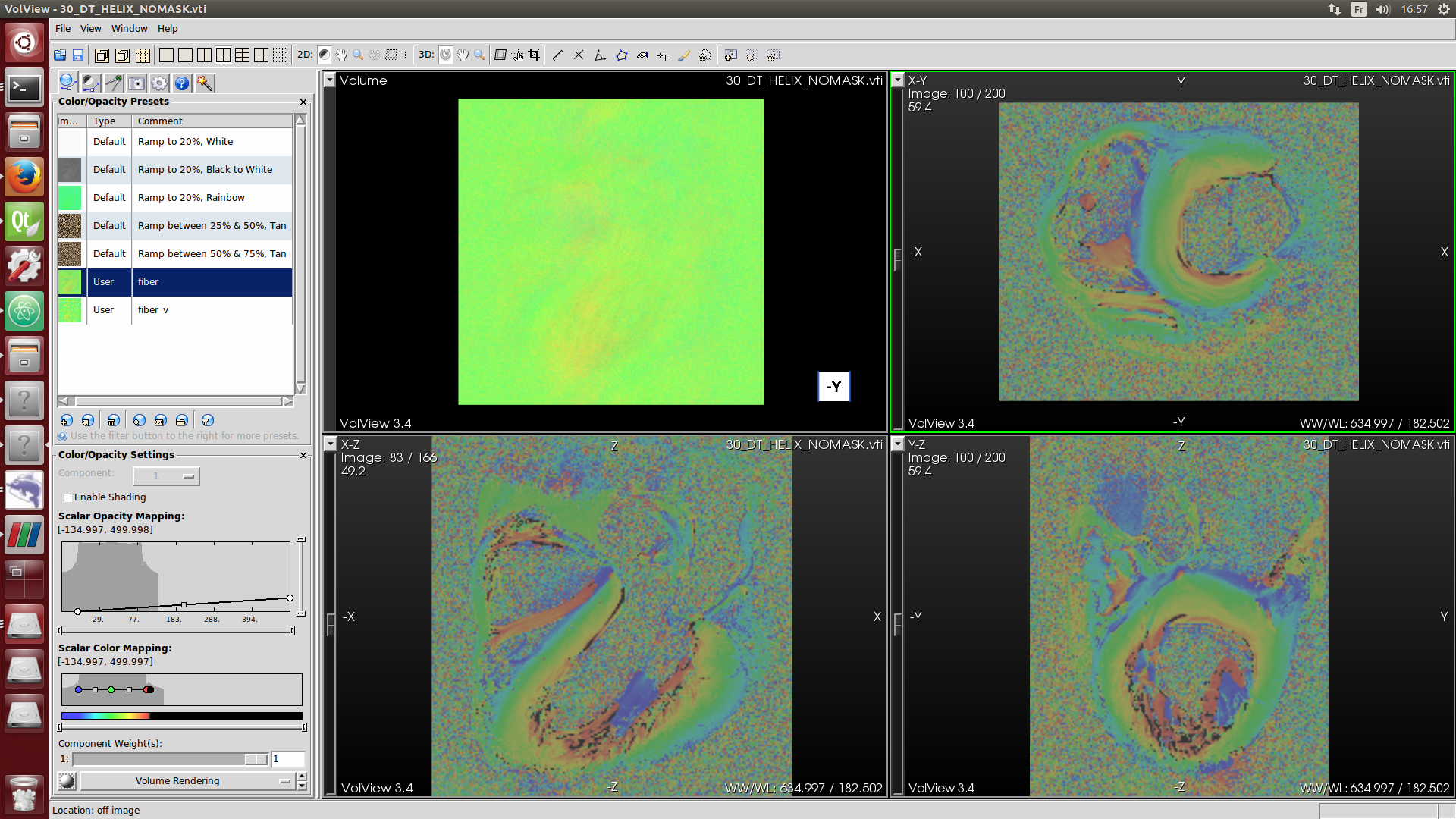
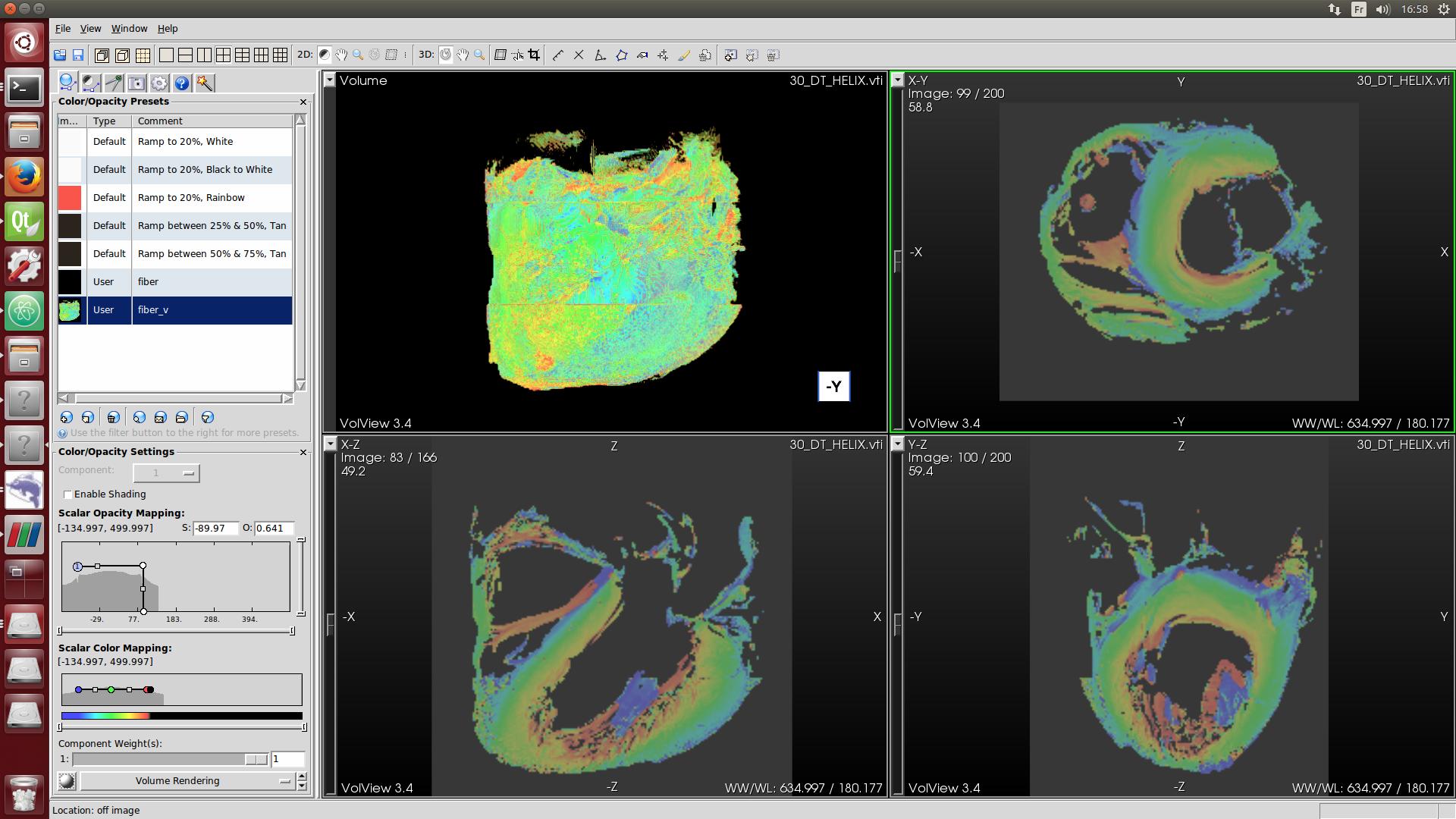
d) Ouverture des vecteurs dans Paraview
#open a terminal
cd
cd Dev/ParaView-4.2.0-Linux-64bit/bin
./paraview
#at the top left-hand corner, clic on file, then open and load the following files: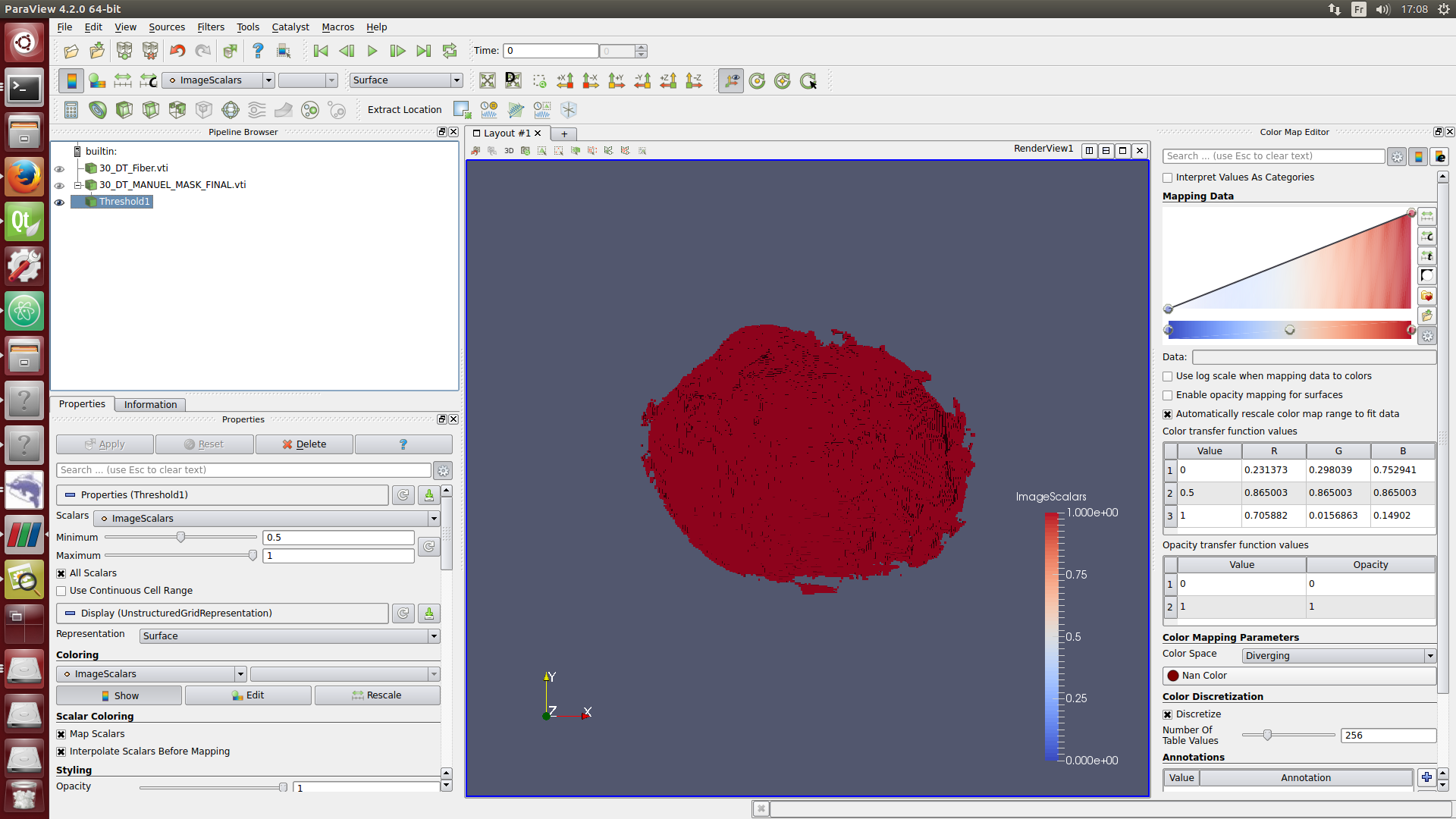
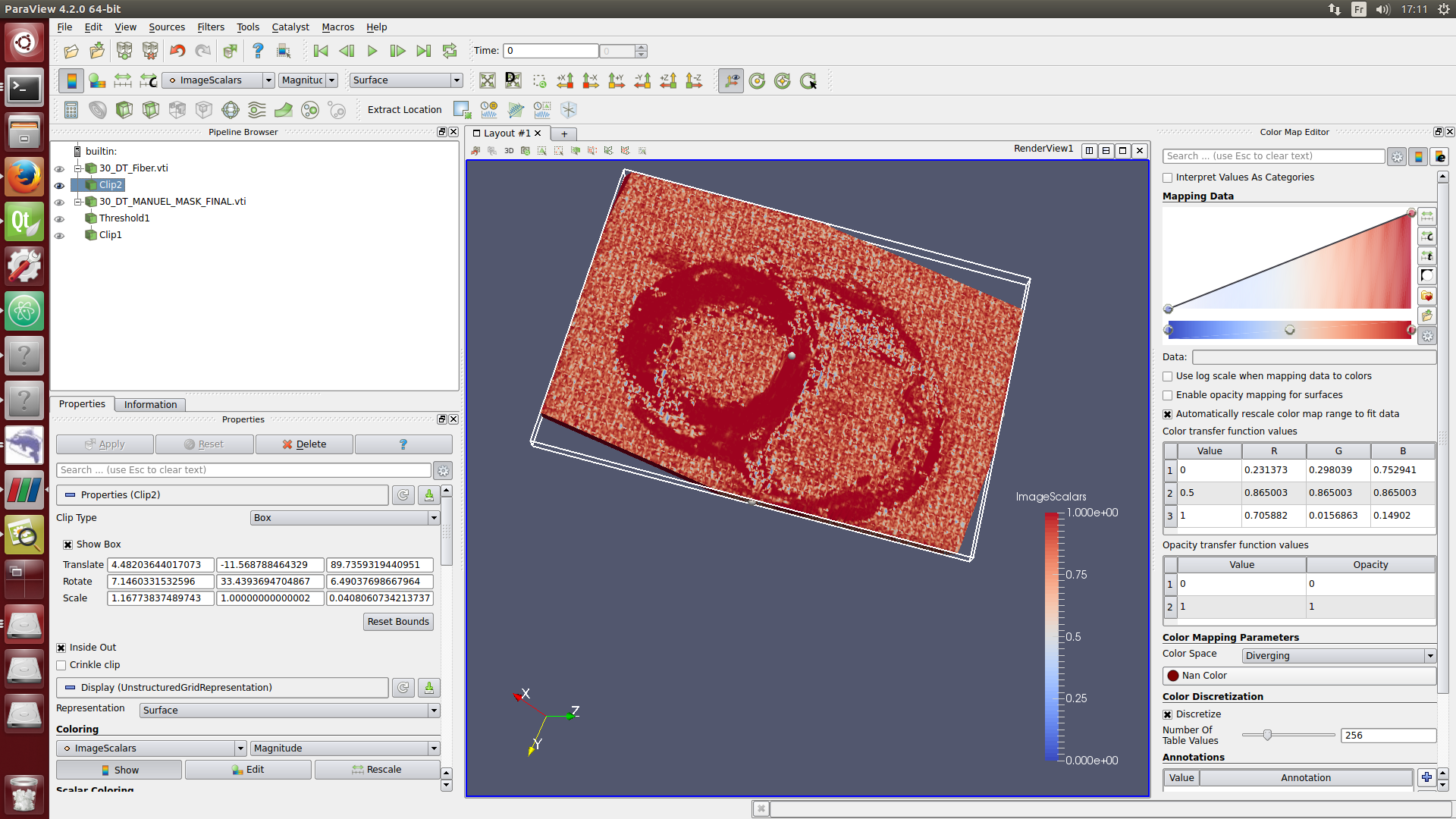
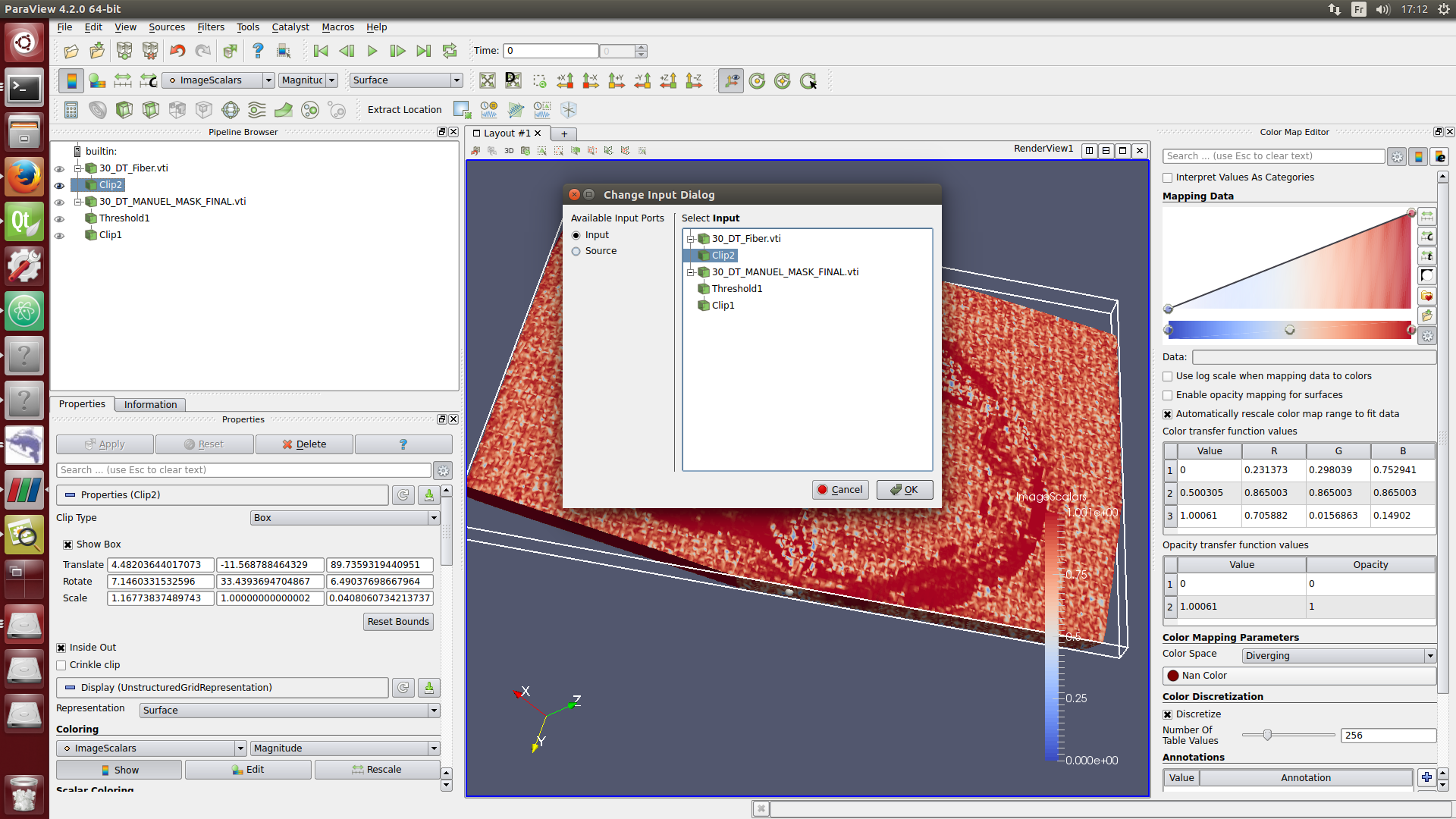
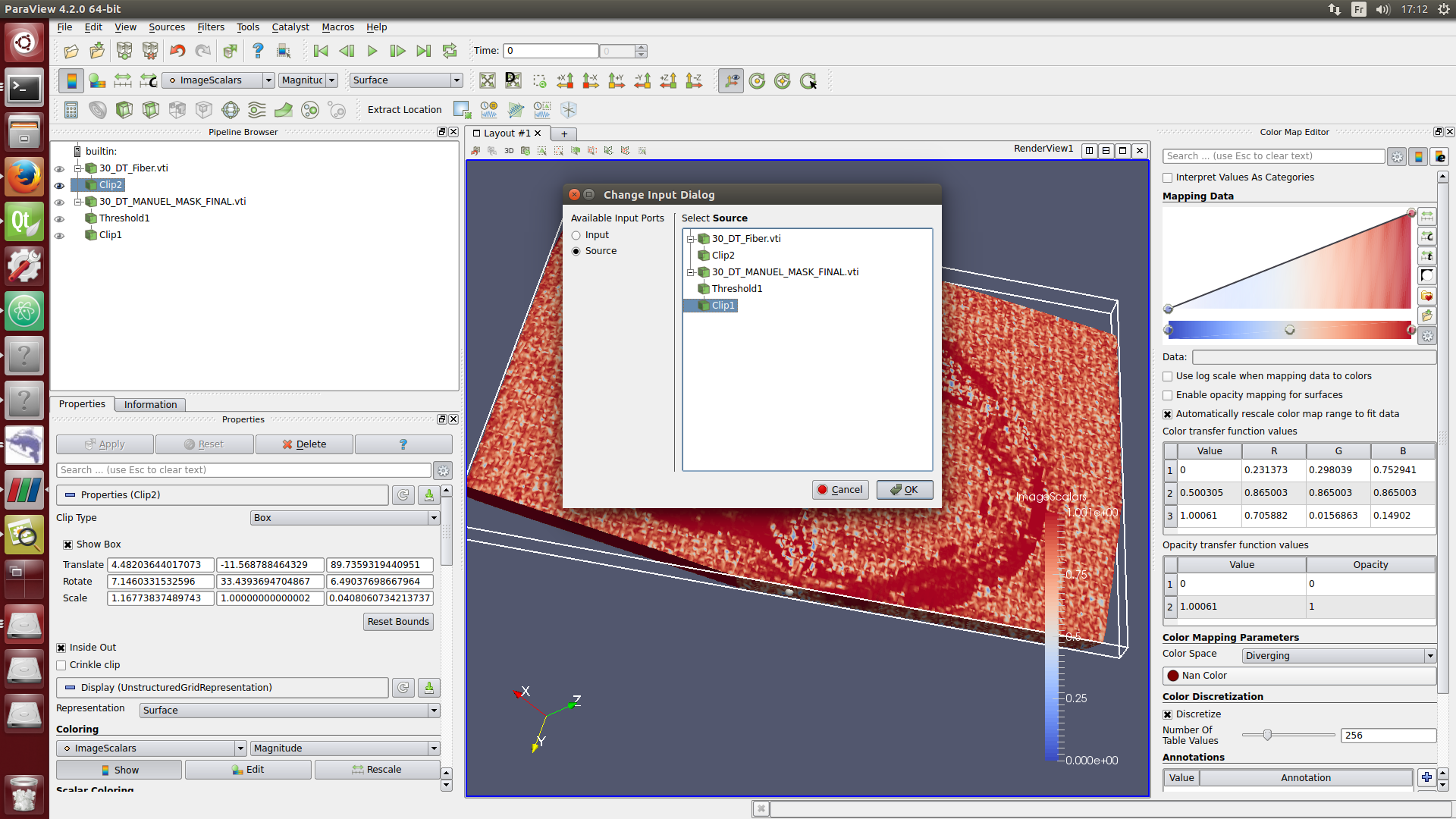
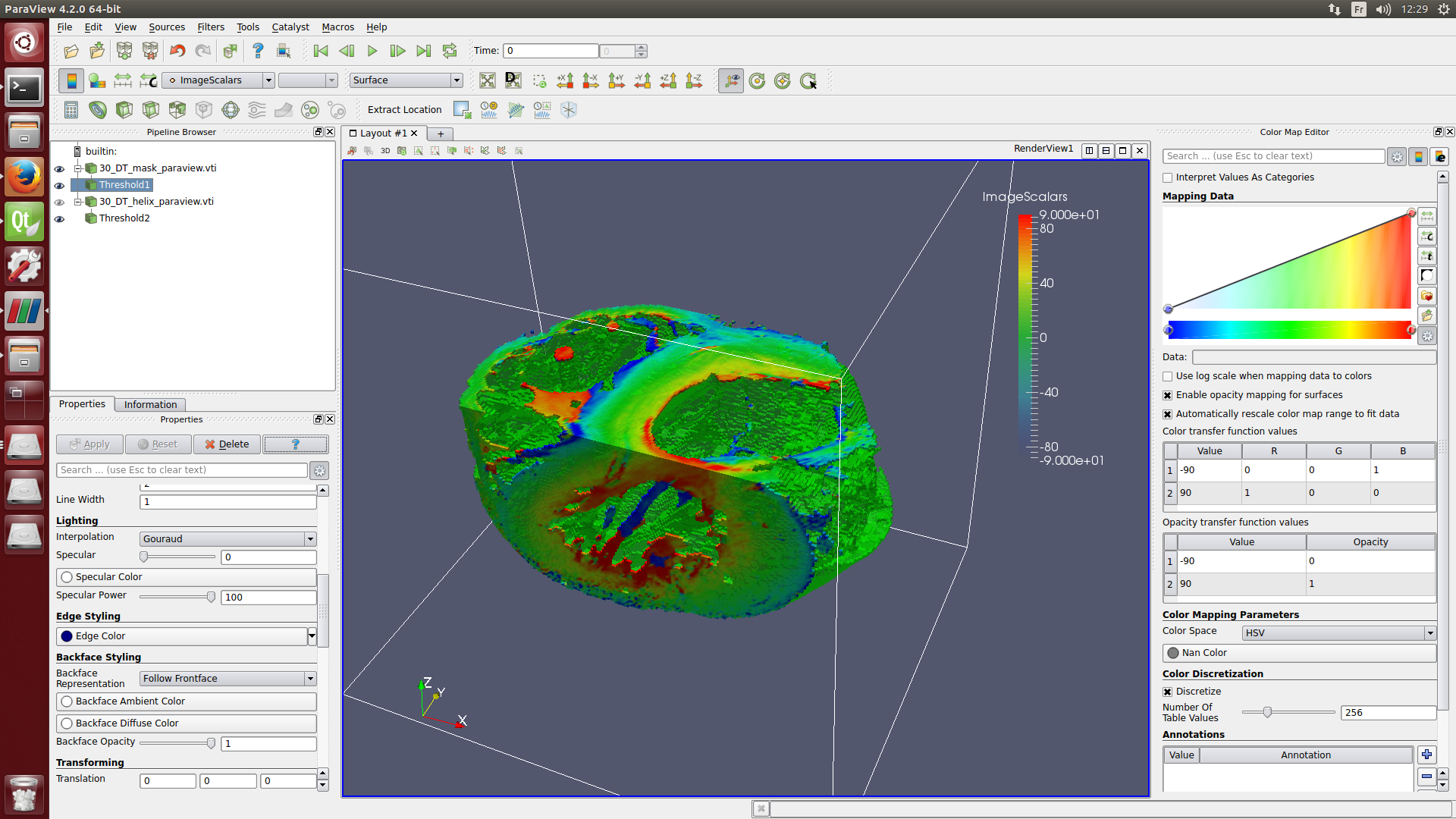
Tutorials/BiventriclesFitting lien
e) Segmentation plus fine du coeur sur ITK-SNAP
Tutorials/Segmentation lien
9) Annexe
9.1) Abréviation
- ANTs : Advanced Normalization Tools
- DTI :
- DWI : diffusion weighted image
- FA : fraction anasotropy
- ITK :
- STI :
- VTK :